Stichwörter: Dynamische Differenzkalorimetrie, monoklonale Antikörper (Monoclonal Antibodies, mAb), Antikörper-Arzneimittelprodukte, kurzzeitige Thermostabilität, hoher Durchsatz, Formulierungsentwicklung
MC178-DE
Abstract
Prüfungen der kurzzeitigen Thermostabilität sind zur Sicherstellung der Produktqualität und mit Blick auf die behördliche Zulassung von Antikörper-Arzneimitteln von entscheidender Bedeutung. Der RS-DSC (Rapid Screening-Differential Scanning Calorimeter) von TA Instruments™ ermöglicht das Screening der Thermostabilität von Pufferformulierungen und Proteinsequenzmodifikationen. Das Gerät stellt eine Plattform für die gleichzeitige Analyse von bis zu 24 Proben in Formulierungskonzentrationen dar. Die Daten in diesem Anwendungshinweis veranschaulichen die Vorteile des RS-DSC von TA Instruments für die Prüfung der kurzzeitigen Thermostabilität von Puffern und die Analyse von Proteinmodifikationen für die Entwicklung von Antikörper-Arzneimitteln.
Einführung
Informationen über den Zusammenhang zwischen der Struktur und der Funktion von Antikörpern sind von grundlegender Bedeutung, wenn es darum geht zu verstehen, wie Funktionsvariationen entstehen, sei es aufgrund von Änderungen der Lösungsbedingungen oder Sequenzmodifikationen. Die Charakterisierung der Antikörperstruktur ist ein wichtiger Schritt zur Analyse dieser Unterschiede. Darüber hinaus ist die Beurteilung der Stabilität von Biomolekülen unter unterschiedlichen thermischen Bedingungen wichtig für die Sicherstellung der Qualität und mit Blick auf die behördliche Zulassung solcher Arzneimittel.
Die dynamische Differenzkalorimetrie (Differential Scanning Calorimetry, DSC) ist eine thermobasierte Technik, die eingesetzt wird, wenn es darum geht, Aufschluss über die Struktur von Antikörpern und die Widerstandsfähigkeit einer Verbindung gegenüber thermischem Stress zu erhalten. Die kurzzeitige Thermostabilität ist ein entscheidendes Qualitätsmerkmal bei der Beurteilung von Antikörper-Arzneimitteln. Tatsächlich handelt es sich um eine zentrale Metrik, die bei der Auswahl und Vorformulierung von Kandidaten bestimmt wird, und um einen wichtigen Parameter bei der Auswahl der Pufferzusammensetzung, zum Beispiel von Hilfsstoffen, Puffersalzen, Detergenzien und dem pH-Wert bei der Entwicklung klinischer Formulierungen. Häufig müssen Dutzende bis Hunderte von Proben untersucht und analysiert werden. Herkömmliche Analysen der dynamischen Differenzkalorimetrie sind zeitaufwändig und erfordern die Untersuchung der Proben einzeln nacheinander sowie eine sorgfältige Reinigung zwischen den Proben, um optimale Ergebnisse zu erzielen. Der RS-DSC von TA Instruments – eine völlig neu konzipierte Plattform für Prüfungen der Thermostabilität mit hohem Durchsatz – ermöglicht die gleichzeitige Analyse von 24 Proben. Durch den Erhalt schneller und umfassender physikalisch-chemischer Daten mittels thermischer Analyse könnten Kosten und Zeitaufwand für die Markteinführung neuer Medikamente reduziert werden.
Thermogramme der dynamischen Differenzkalorimetrie für Antikörper sind bemerkenswert vielfältig. Manche Antikörper ergeben einen einzelnen Peak, während andere mehrere unterschiedliche Peaks oder überlappende Peaks aufweisen, die sich als Schultern an einem größeren Peak darstellen. Die Variabilität dieser Thermogramme könnte mit der Flexibilität der Gelenkregion zusammenhängen [1]. Untersuchungen an Multidomänenproteinen deuten darauf hin, dass diese komplexen und überlappenden Peaks auf Wechselwirkungen zwischen Domänen zurückzuführen sein könnten [2]. In erweiterten Studien zur dynamischen Differenzkalorimetrie wurde sogar erfolgreich die Entfaltungstemperatur einzelner Domänen innerhalb der schweren Kette von Antikörpern bestimmt [3]. Bei solchen Studien besteht das Ziel letztendlich darin, die Thermostabilität zu ermitteln, da eine unzureichende Stabilität die Löslichkeit erheblich beeinträchtigen und zur Aggregation führen kann – ein häufiges Problem bei Biotherapeutika.

Experimente und Methoden
Es wurde unter Einhaltung der Packungsanleitung eine Zubereitung von Herceptin™ Trastuzumab in Histidin-Formulierungspuffer (18,4 mg/ml Trehalose-Dihydrat, 0,08 mg/ml Polysorbat 20, 0,49 mg/ml Histidin-HCl, 0,32 mg/ml Histidin, pH 6,0) mit 21 mg/ml hergestellt und bis zur Verwendung bei 4 °C aufbewahrt. Für die Analyse in verschiedenen Puffern wurde unter Verwendung eines Amicon™ Ultra-Zentrifugenfilters mit 10 kDa Molekulargewichtsausschluss ein Pufferaustausch der Antikörperzubereitung durchgeführt.
Der Boratpuffer bestand aus 50 mM Borsäure, 50 mM NaCl, 2 mM EDTA bei pH 8,0. Phosphatgepufferte Salzlösung, ein gängiger Arbeitspuffer, bestand aus Gibco™ DPBS mit 1 mM EDTA bei pH 7,4. Der für KADCYLA™, ein auf dem Trastuzumab-Antikörpergerüst aufgebautes Antikörper-Wirkstoff-Konjugat, verwendete Succinat-Formulierungspuffer bestand aus 10 mM Natriumsuccinat, 6 % w/v Saccharose, pH 5,0.
Die Antikörperproben für das Mutationsscreening stammten von Cell Signaling Technology und waren für die Analyse in PBS (pH 7,4), 20 mg/ml, formuliert.
Die Proteinprobe wird direkt in die Einweg-Mikrofluidikchips (Microfluidic Chips, MFCs) aus Glas pipettiert, deren Kanal 11 μl flüssige Probe aufnehmen kann. Nach Zugabe der Proteinlösung werden die Mikrofluidikchips mit selbstklebenden Glasdeckgläsern verschlossen, um ein Verdunsten der Probe während des Erhitzens auf 100 °C zu vermeiden. Die fertigen Einweg-Mikrofluidikchips werden auf der Probenseite jedes der beiden Kalorimeter im RS-DSC von TA Instruments platziert (Abb. 1). Ein wiederverwendbarer Polyetheretherketon-Chip befindet sich auf der Referenzseite. Es können bis zu 24 Proben gleichzeitig in einem Temperaturbereich von 20 °C bis 100 °C und mit voreingestellten Scanraten von 1 °C/min oder 2 °C/min analysiert werden. Da die Reinigung zwischen den Scans entfällt, können an einem typischen Arbeitstag bis zu 96 Proben gemessen werden.
Der RS-DSC von TA Instruments wurde mit der Software RS-DSCRun verwendet und konnte sich vor Beginn jedes Scans 1800 s lang auf die Anfangstemperatur äquilibrieren. Jede Probe wurde dreimal gemessen und mit 1 °C/min oder 2 °C/min über den gesamten Temperaturbereich (20 °C bis 100 °C) gescannt.
Zur Sicherstellung der Genauigkeit aller 24 Kalorimeter wurde das Gerät zunächst mit Dipalmitoylphosphatidylcholin kalibriert und validiert (akzeptable Tmax-Verschiebung von weniger als 0,2 °C vom erwarteten Literaturwert) [4].
Die Datenverarbeitung erfolgte mit der Software NanoAnalyze™ (v4.0.0). Neue Funktionen in der Software NanoAnalyze ermöglichen die automatische Erkennung des mittleren Punkts der Denaturierungstemperatur (Tmax) und den Vergleich von bis zu 96 Thermogrammen. Die Software NanoAnalyze v4.0.0 enthält einen neuen Softwarealgorithmus, RapidDSC, der Tmax in Thermogrammen der dynamischen Differenzkalorimetrie automatisch erkennt, indem er im angegebenen Datenbereich nach Peaks sucht. Auf den Peak wird eine automatische Grundlinie angewendet und zur Verbesserung der Genauigkeit bei der Bestimmung der Tmax-Temperatur verwendet. Automatische Peakerkennungsfunktionen zeigen bis zu drei Peaks im relevanten Temperaturbereich an. Die Automatisierungsfunktionen lassen sich im Popup-Fenster „Baseline and Tmax Editor“ einfach verändern, um das Erkennungsfenster einzugrenzen, einen spezifischen Bereich für die Peakidentifizierung auszuwählen oder die automatisierte Grundlinie und Tmax anzupassen. Die Daten aus der Tmax-Automatisierung können zu Vergleichszwecken als Tabelle exportiert oder in den Registerkarten in RapidDSC zur Anzeige und Überlagerung der Tmax-Daten dargestellt werden.
Zum Vergleich mit der mit RapidDSC ermittelten automatisierten Tmax wurde eine manuelle Analyse in der NanoAnalyze-Software durchgeführt. Für den manuellen Vergleich mit den automatischen Peakerkennungsfunktionen wurde jeder einzelne Scan unter Verwendung der Mole an Protein im aktiven Zellenvolumen auf die molare Wärmekapazität normalisiert. Als Grundlinie wurde auf die normalisierten Daten ein Polynom vierter oder fünfter Ordnung angewendet. Die Tmax wurde visuell geschätzt und mit der automatisierten Tmax-Ausgabe für jeden Übergang verglichen. Durch Anpassen an Modelle, wie in „NanoDSC: What to Consider when Choosing a Baseline and Model“ beschrieben, ist eine weitere Analyse der Thermogramme möglich [5]. Für die Analyse der Antikörperstabilität wird die Verwendung von Voigt-Modellen empfohlen.
Ergebnisse und Diskussion
Formulierungspuffer-Screening
Die Thermostabilität ist ein entscheidendes Qualitätsmerkmal eines biologischen Arzneimittels. Die dynamische Differenzkalorimetrie ist das wichtigste Hilfsmittel zur Charakterisierung der Auswirkung der Lösungsumgebung auf die Proteinstabilität. Die Proteinstabilität wird durch eine Reihe von Variablen in der Lösungsumgebung beeinflusst, wie z. B. durch den pH-Wert, den Puffertyp, die Ionenstärke, vorhandene Hilfsstoffe und Detergenzien. Diese Effekte machen sich in Form einer kleinen Verschiebung der Tmax oder als größere Änderungen von bis zu mehreren zehn Grad bemerkbar [6]. Der RS-DSC von TA Instruments mit parallelem Durchsatz wurde speziell mit Blick auf die Anforderungen in der biopharmazeutischen Industrie entwickelt und erleichtert das Screening von Lösungsbedingungen für biologische Proteinarzneimittel, sodass im Vergleich zu herkömmlichen mikrokalorimetrischen Methoden
schneller Entscheidungen getroffen werden können.
Um zu zeigen, wie die Daten aus dem Formulierungsscreening bei der Auswahl von Pufferkomponenten helfen können, haben wir den Antikörper Herceptin Trastuzumab in Dreifachbestimmungen unter vier gängigen Pufferbedingungen analysiert (Abb. 2 und Tabelle 1). Für diese Studie wurden Antikörper in verschiedenen Formulierungspuffern bei ~ 20 mg/ml gescreent. Der RS-DSC von TA Instruments ist jedoch speziell für die Analyse von Proteinkonzentrationen in Formulierungsstärke von bis zu 330 mg/ml und darüber ausgelegt und ermöglicht daher die Prüfung der Thermostabilität unverdünnter Arzneimittel. Weitere Informationen gibt es im Anwendungshinweis „Rapid Thermal Stability Screening of High Concentration Biologic Drugs“ (Schnelles Screening der Thermostabilität von hochkonzentrierten biologischen Arneimitteln) [7].
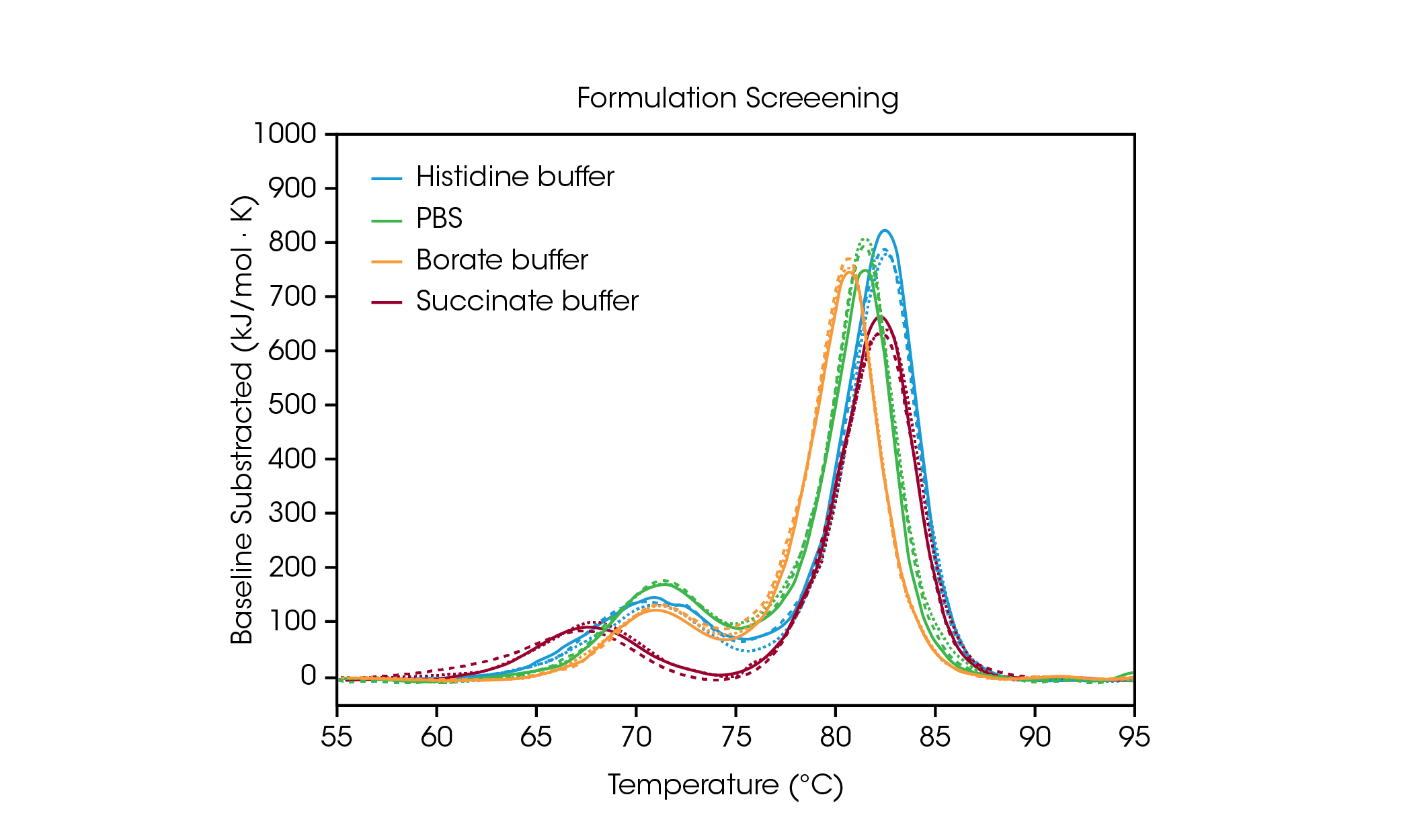
Tabelle 1. Übergangstemperaturen für das Formulierungsscreening Mittelwert ± SD, n = 3.
| Puffer | Tmax,1 (°C) | Tmax,2 (°C) |
|---|---|---|
| Histidin | 70,76 ± 0,38 | 82,66 ± 0,09 |
| Borat | 70,84 ± 0,08 | 80,69 ± 0,02 |
| PBS | 71,12 ± 0,06 | 81,48 ± 0,02 |
| Succinat | 67,71 ± 0,22 | 82,66 ± 0,08 |
Das erste Auffaltungsereignis, die Auffaltung der Domäne CH2, wird durch die Histidin-, Borat- oder PBS-Puffer nicht wesentlich beeinflusst [8]. Allerdings destabilisiert der Succinatpuffer die Domäne CH2, was zu einer Verringerung der Temperatur zum Beginn der Auffaltung und der Tmax,1 um ca. 3 °C führt. Was den Hauptübergang anbelangt, die Fab- und H3-Auffaltungsereignisse, wirken der Histidin- und der Succinatpuffer mit einer Tmax,2 von 82,66 °C am stärksten stabilisierend. Der Hauptübergang ist in Boratpuffer am wenigsten stabil bei einer Tmax,2 von 80,69 °C. Wenig überraschend ist die Pufferformulierung mit der stärksten stabilisierenden Wirkung für Trastuzumab in diesem Probensatz der Histidinpuffer, der auch für die fertige Darreichungsform des zugelassenen Arzneimittels verwendet wird.
–>Die automatisierte Tmax-Erkennung in der Software NanoAnalyze ermöglicht die genaue Identifizierung von bis zu drei Übergängen in Proteinstabilitätstests und erleichtert damit die Analyse der Daten aus einem Formulierungsscreening. Um die Genauigkeit des Peakauswahl-Algorithmus zu demonstrieren, werden die automatischen Tmax,1 und Tmax,2 für Trastuzumab bei 50 mg/ml in Histidin-Formulierungspuffer mit denen bei manueller Peakauswahl verglichen (Abb. 3 und Tabelle 2). Bei der automatisierten Analyse wurden nach Anwendung eines Basispolynoms vierter Ordnung mithilfe der Auto-Startup-Hook-Erkennungsfunktion, die keine Angabe des interessierenden Temperaturbereichs erfordert, zwei Peaks erkannt.
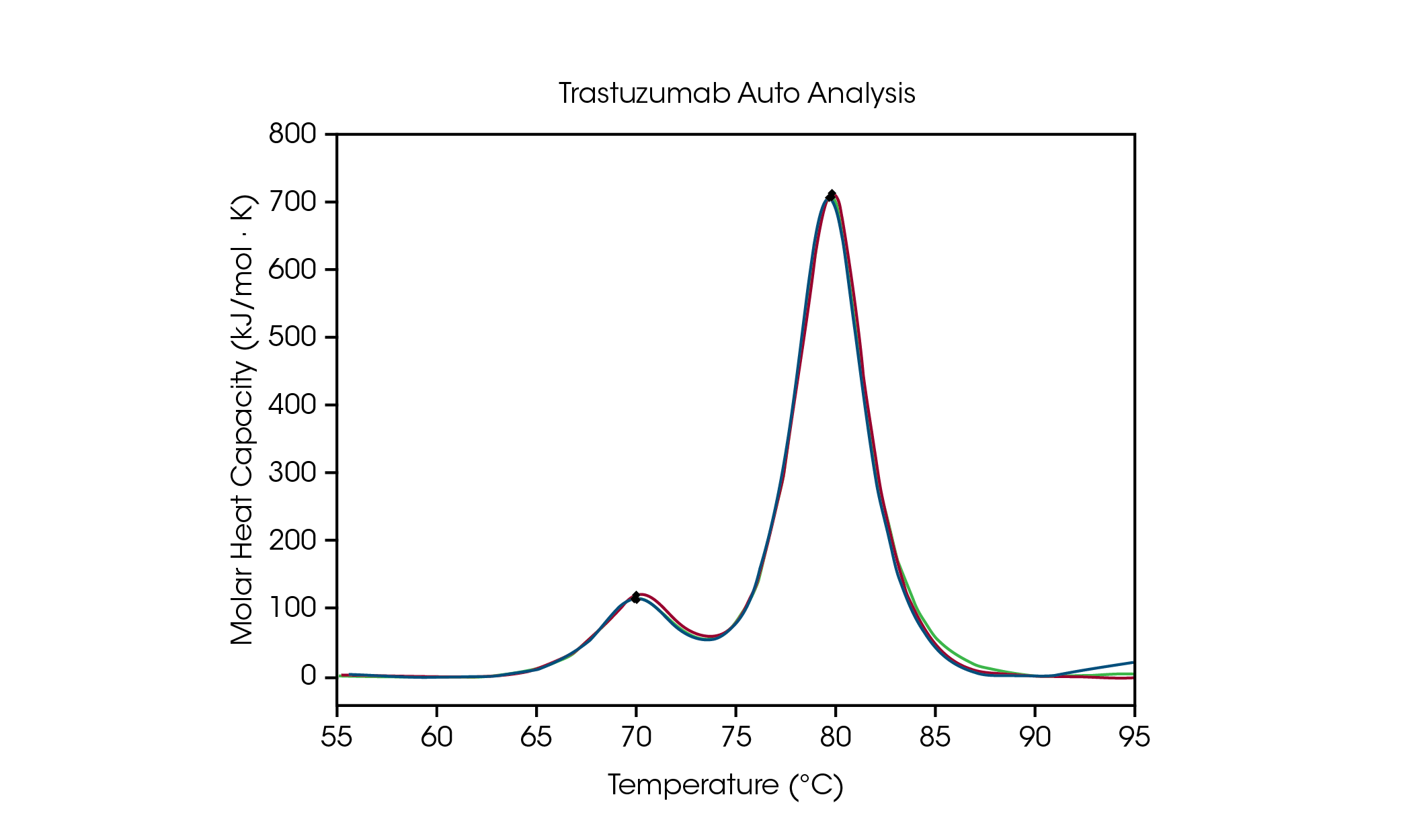
Tabelle 2. Vergleich zwischen automatisierter und manueller Tmax-Erkennung mit Trastuzumab
| Probe | Auto Tmax,1 (°C) | Manuelle Tmax,1 (°C) | Auto Tmax,2 (°C) | Manuelle Tmax,2 (°C) |
|---|---|---|---|---|
| Replikat 1 | 71,93 | 72,04 | 82,92 | 82,88 |
| Replikat 2 | 71,81 | 71,93 | 82,76 | 82,75 |
| Replikat 3 | 71,93 | 71,97 | 82,90 | 82,83 |
| Durchschnitt | 71,89 ± 0,07 | 71,98 ± 0,06 | 82,86 ± 0,09 | 82,82 ± 0,07 |
Die Auffaltung der Domäne CH2 hat bei der automatisierten Peakanalyse eine Tmax,1 von 71,89 °C verglichen mit 71,98 °C bei der manuellen Peakanalyse. Der Hauptübergang bei der automatisierten Analyse erfolgt bei einer Tmax,2 von 82,86 °C gegenüber 82,82 °C bei der manuellen Peak-Analyse. Die Reproduzierbarkeit bei Trastuzumab ist hervorragend. Bei der Analyse von Dreifachproben liegt die Tmax beider Peaks innerhalb der Genauigkeitsgrenzen des Geräts von ± 0,2 °C, sowohl bei der manuellen als auch bei der automatisierten Peakanalyse. Zur Komplettierung der Datenauswertung wird immer auch eine Sichtprüfung empfohlen. Dieser Datensatz zeigt, dass die automatisierten Analysefunktionen der dynamischen Differenzkalorimetrie eine genaue Erkennung der max in Thermogrammen mit mehreren Übergängen ermöglichen.
Screening von Sequenzmodifikationen
Proteinmutationen sind eine gängige Methode zur Optimierung der Proteinstruktur und -funktion, und selbst Modifikationen einzelner Aminosäuren können einen messbaren Einfluss auf die Gesamtstabilität des Proteins haben. Die dynamische Differenzkalorimetrie kann Aufschluss über den Einfluss einer Mutation auf die Thermostabilität eines Proteins geben und somit bei der Entscheidungsfindung in der Entwicklungspipeline für biologische Arzneimittel hilfreich sein. Um die Auswirkungen einer Sequenzmodifikation auf die Stabilität zu demonstrieren, wurde eine kleine Gruppe gentechnisch veränderter Antikörper auf Veränderungen der temperaturabhängigen Auffaltung infolge einzelner Aminosäuremutationen in der Proteinsequenz untersucht (Abb. 4 und Tabelle 3).
Im ursprünglichen Antikörper findet die Auffaltung der Domänen CH2, Fab und CH3 innerhalb eines thermischen Hauptübergangs bei einer Tmax von 75,92 °C statt. Ausgehend von dem ursprünglichen Protein wurde eine einzelne Aminosäuremutation hergestellt, die mit einer Tmax von 75,52 °C keinen wesentlichen Einfluss auf die kurzzeitige Thermostabilität hatte (Mutation 1). Zusätzliche einzelne Aminosäuremutationen haben allerdings erhebliche Auswirkungen auf die Stabilität des Proteins (Mutation 2 und Mutation 3). Bei Mutation 2 ist eine erkennbare Schulter zu beobachten, wahrscheinlich das Resultat einer Destabilisierung in der Domäne CHH2 mit einer zusätzlichen Tmax,1 von 70,04 °C, verbunden mit einer kleinen Verschiebung des Hauptübergangs relativ zum Ausgangsprotein mit einer Tmax,2 von 75,14 °C. Mutation 3 führt zu einer erheblichen Destabilisierung der Domäne CH2 mit einem vollständigen, zusätzlichen Auffaltungspeak mit einer Tmax,1 von 64,85 °C. Der Hauptübergang bleibt mit einer Tmax,2 von 75,29 °C relativ unbeeinflusst. Wie die erhebliche Destabilisierung von Mutation 3 zeigt, hat eine Modifikation nicht immer die gleiche Wirkung; eine Rolle spielen sowohl der Ort der Modifikation als auch die physikalisch-chemischen Eigenschaften der neuen Aminosäure. Die Optimierung der gewünschten funktionellen Vorteile der Sequenzmodifikation über die strukturelle Stabilität des Proteins trägt zum Verständnis der Struktur-Funktions-Beziehung bei und kann der Entwicklung neuartiger Therapeutika erleichtern.
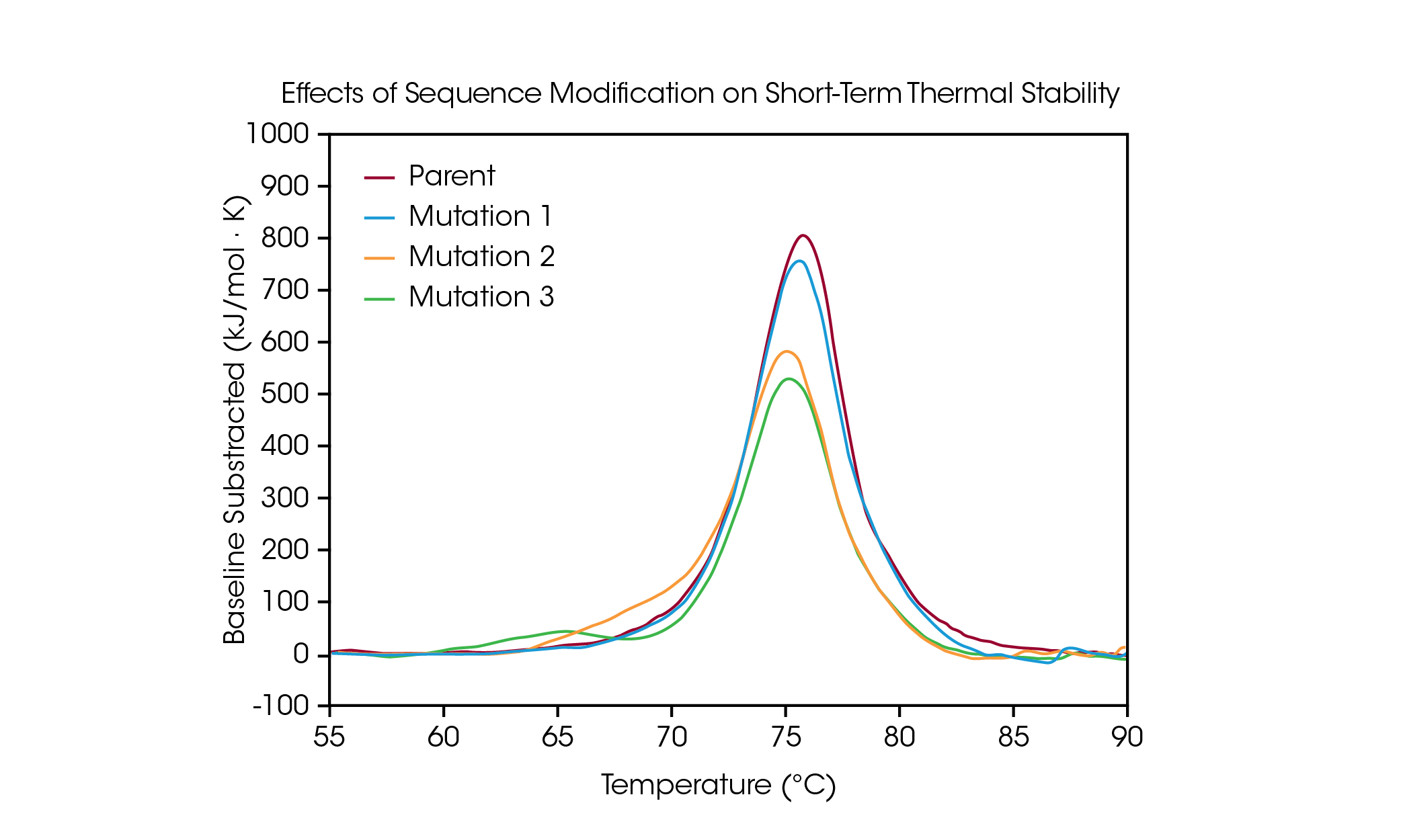
Tabelle 3. Übergangstemperaturen im Mutationsscreening Mittelwert ± SD, n = 3. *Bestimmt mit dem Voigt-Modell.
| Probe | Tmax,1 (°C) | Tmax,2 (°C) |
|---|---|---|
| Ausgangsmolekül | — | 75,92 ± 0,15 |
| Mutation 1 | — | 75,52 ± 0,14 |
| Mutation 2 | 70,04 ± 0,86* | 75,14 ± 0,06 |
| Mutation 3 | 64,85 ± 0,05* | 75,29 ± 0,06 |
Fazit
Das Screening der Auswirkungen der Lösungsumgebung und der Sequenzmodifikation auf die kurzzeitige Thermostabilität ist für die Entwicklung hochwertiger pharmazeutischer Arzneimittel von entscheidender Bedeutung. Der RS-DSC von TA Instruments ermöglicht ein Hochdurchsatz-Screening der Thermostabilität durch gleichzeitiges Testen von bis zu 24 Proben und beschleunigt damit die Entscheidungsfindung erheblich. Wir zeigen hier eine genaue und reproduzierbare Analyse von Antikörperproben mit komplexem multiplem Übergang und die schnelle Bestimmung der Auswirkungen von Pufferkomponenten und Mutationen auf die gesamte Proteinstruktur. Neue Algorithmen in der Datenverarbeitungssoftware NanoAnalyze erleichtern die Analyse der großen Mengen von thermischen Daten aus dem Hochdurchsatz-Screening. Insgesamt stellt der RS-DSC von TA Instruments eine neue Plattform zur Charakterisierung der Stabilität von Antikörper-Arzneimittelprodukten dar – einer wichtigen Variable zum Verständnis der Widerstandsfähigkeit gegenüber thermischem Stress und der Produktqualität und als Daten für die behördliche Zulassung.
Literatur
- V. T. Oi, T. M. Vuong, R. Hardy, J. Reidler, J. Dangl, L. A. Herzenberg und L. Stryer, „Correlation between segmental flexibility and effector function of antibodies“, Nature, Vol. 307, S. 136-140, 1984.
- P. L. Privalov und S. A. Potekhin, „Scanning microcalorimetry in studying temperature-induced changes in proteins“, Methods in Enzymology, Vol. 131, S. 5-51, 1986.
- J. Wen, Y. Jiang und L. Nahri, „Effect of Carbohydrate on Thermal Stability of Antibodies“, American Pharmaceutical Review, Vol. 11, S. 98, 2008.
- „Standard Practice for Calibration of Fixed-Cell Differential Scanning Calorimeters“, ASTM E2603-15, 2023.
- „NanoDSC: What to Consider when Choosing a Baseline and Model“, TA Instruments, MC154.
- C. M. Johnson, „Differential scanning calorimetry as a tool for protein folding and stability“,Archives of Biochemistry and Biophysics, Vol. 531, S. 100-109, 2013.
- „Rapid Thermal Stability Screening of High Concentration Biologic Drugs“, TA Instruments, MC177.
- K. J. Arlotta, A. V. Gandhi, H.-N. Chen, C. S. Nervig, J. F. Carpenter und S. C. Owen, „In-Depth Comparison of Lysine-Based Antibody-Drug Conjugates Prepared on Solid Support Versus in Solution“, Antibodies, Vol. 7, S. 6, 2018.
Hier klicken, um eine Druckversion dieses Anwendungshinweises herunterzuladen.

