Palabras clave: calorimetría diferencial de barrido, anticuerpos monoclonales (Acm), medicamentos con anticuerpos, estabilidad térmica a corto plazo, alto rendimiento, desarrollo de formulación
MC178-ES
Resumen
Las pruebas de estabilidad térmica a corto plazo son cruciales para asegurar la calidad del producto y dar soporte a la aprobación regulatoria de los medicamentos con anticuerpos. El RS-DSC (Calorímetro Diferencial de Barrido de Escaneo Rápido [Rapid Screening-Differential Scanning Calorimeter, RS-DSC]) ofrece escaneo de estabilidad térmica de formulaciones de amortiguador y modificaciones de secuencia de proteína. El instrumento proporciona una plataforma para el análisis simultáneo de hasta 24 muestras a concentraciones de potencia de formulación. En esta nota de aplicación se proporcionan datos que ilustran la utilidad del RS-DSC de TA Instruments para permitir el escaneo de estabilidad térmica a corto plazo de amortiguadores y el análisis de modificaciones de proteína para el desarrollo de medicamentos con anticuerpos.
Introducción
El entendimiento de la relación estructura-función de los anticuerpos es fundamental para comprender cómo surgen variaciones funcionales, ya sea por alteraciones de las condiciones de la solución o modificaciones de secuencia. La caracterización de la estructura de los anticuerpos es un paso importante en la delineación de estas diferencias. Además, la evaluación de la estabilidad de las biomoléculas en diferentes condiciones térmicas es crucial para asegurar la calidad y la aprobación regulatoria de estos medicamentos.La Calorimetría Diferencial de Barrido (
Differential Scanning Calorimetry, DSC) es una técnica térmica que se emplea para comprender la estructura de los anticuerpos y evaluar la resistencia de un compuesto al estrés térmico. La estabilidad térmica a corto plazo es un atributo de calidad fundamental en la evaluación de medicamentos con anticuerpos. Es una medida clave que se emplea en la selección de candidatos y la preformulación, y es un parámetro importante en la selección de la composición del amortiguador, incluso excipientes, sales amortiguadoras, detergentes y pH en el desarrollo de formulaciones clínicas. Debido a la naturaleza de este escaneo, es común tener entre decenas y cientos de muestras que se deben procesar y analizar. Los estudios de DSC tradicionales son lentos, solo pueden analizar una muestra a la vez y requieren una limpieza meticulosa entre muestras para obtener mejores resultados. El RS-DSC de TA Instruments, una nueva plataforma para pruebas de estabilidad térmica de alto rendimiento, redefine el panorama de las pruebas de estabilidad térmica al analizar simultáneamente 24 muestras. Proporcionar información fisicoquímica rápida e integral mediante el análisis térmico reduciría el costo y el tiempo necesarios para llevar nuevos fármacos al mercado.
Los termogramas de DSC para anticuerpos son notablemente diversos. Algunos anticuerpos muestran un solo pico, mientras que otros muestran múltiples picos separados o picos superpuestos que se manifiestan como hombros sobre un pico desplegado más grande. Se ha sugerido que la variedad de estos termogramas se correlaciona con la flexibilidad de la región de bisagra [1]. Estudios sobre proteínas multidominio indican que estos picos complejos y superpuestos pueden ser el resultado de interacciones interdominio [2]. En estudios de DSC avanzados incluso se ha precisado con éxito la temperatura de despliegue de dominios individuales dentro de la porción de la cadena pesada de los anticuerpos [3]. El objetivo final de estos estudios es verificar la estabilidad térmica, porque una estabilidad deficiente puede tener repercusiones significativas sobre la solubilidad, lo que lleva a agregación – un problema común en bioterapéutica.

Experimentos y métodos
Herceptin™ Trastuzumab se preparó según las instrucciones del paquete en amortiguador de formulación de histidina (18.4 mg/ml de dihidrato de trehalosa, 0.08 mg/ml de polisorbato 20, 0.49 mg/ml de histidina HCl, 0.32 mg/ml de histidina, pH 6.0) a 21 mg/ml y se almacenó a 4 °C hasta su uso. Para el análisis en diferentes amortiguadores, el anticuerpo se intercambió con un amortiguador al utilizar un filtro para centrifugado de corte de 10 kDa de peso molecular Amicon™ Ultra.
El amortiguador de borato estuvo compuesto de ácido bórico 50 mM, NaCl 50 mM y EDTA 2 mM a pH 8.0. El PBS, un amortiguador de trabajo común, estuvo compuesto de DPBS Gibco™ con EDTA 1 mM a pH 7.4. El amortiguador de formulación de succinato utilizado para KADCYLA™, un conjugado de fármaco-anticuerpo construido sobre la estructura del anticuerpo trastuzumab, estuvo compuesto de succinato de sodio 10 mM, sacarosa al 6 % p/v, pH 5.0.
Cell Signaling Technology diseñó y suministró las muestras de anticuerpos para el escaneo de mutaciones, y esas muestras se formularon en PBS (pH 7.4) a 20 mg/ml para análisis.
La muestra de proteína se pipetea directamente hacia los chips microfluídicos (CMF) de vidrio desechables con un canal diseñado para contener 11 μl de muestra líquida. Después de agregar la solución de proteína, los chips microfluídicos se sellan con cubreobjetos de vidrio con adhesivo en el reverso para contener la muestra durante el calentamiento a 100 °C. Los MFC ensamblados se colocan en el lado de la muestra de cada calorímetro gemelo en el RS-DSC de TA Instruments (figura 1). En el lado de referencia queda un chip de polieteretercetona (PEEC) reutilizable. Se pueden analizar hasta 24 muestras simultáneamente con un rango de temperatura de 20 a 100 °C y velocidades de escaneo preestablecidas de 1 o 2 °C/min. Al eliminar la necesidad de limpieza entre escaneos, se pueden procesar hasta 96 muestras en un día laboral típico.
El RS-DSC de TA Instruments, operado con el software RSDSCRun, se configuró para equilibrarse a la temperatura inicial durante 1800 segundos antes del inicio de cada escaneo. Cada muestra se procesó por triplicado y se escaneó a 1 o 2 °C/min en todo el rango de temperatura (20 a 100 °C).
Para asegurar la precisión en los 24 calorímetros, el instrumento se calibró y validó primero con dipalmitoilfosfatidilcolina (DPFC), con un cambio de Tmáx aceptado de menos de 0.2 °C desde el valor en la literatura esperado[4].
Los datos se procesaron con el uso del Software NanoAnalyze™ (v4.0.0). Las nuevas funciones del software NanoAnalyze permiten la detección automatizada de la temperatura del punto medio de desnaturalización (Tmáx) y la comparación de hasta 96 termogramas. El Software NanoAnalyze v4.0.0 incluye un nuevo algoritmo de software, RapidDSC, que detectará automáticamente Tmáx en termogramas de DSC al buscar picos en el rango de datos especificado. Se aplica una basal automática en el pico y se utiliza para refinar más la temperatura Tmax a fin de mejorar la precisión. Las funciones de detección automática de picos muestran hasta tres picos en el rango de temperatura de interés. Las funciones de automatización se pueden editar con facilidad en la ventana emergente “Editor de basal y de Tmáx” (“Baseline and Tmáx Editor”) para reducir la ventana de detección, seleccionar un rango específico para la identificación de picos o ajustar la basal y la Tmáx automatizadas. Los datos tabulados de la automatización de Tmáx se pueden exportar para una comparación fácil, o se pueden ver en las pestañas de visualización y superposición de Tmáx en RapidDSC.
Se realizó análisis manual en el software NanoAnalyze para comparación con la Tmáx automatizada detectada al usar RapidDSC. Para comparación manual con las funciones de detección de pico automatizadas, cada escaneo individual se normalizó a la capacidad calorífica molar al utilizar los moles de proteína en el volumen celular activo. Se aplicó un polinomio de cuarto o quinto orden como la basal para los datos normalizados. La Tmáx se estimó visualmente y se comparó con la salida de Tmáx automatizada para cada transición. El análisis adicional de los termogramas puede completarse mediante integración con modelos como se describe en “NanoDSC: qué considerar cuando se elige una basal y un modelo” (“NanoDSC: What to Consider when Choosing a Baseline and Model“) [5]. Para el análisis de la estabilidad de los anticuerpos se recomienda el uso de los modelos de Voigt.
Resultados y discusión
Escaneo de formulación de amortiguador
La estabilidad térmica es un atributo de calidad crucial de un medicamento biológico. La DSC es una herramienta primaria implementada para caracterizar el efecto del entorno de la solución sobre la estabilidad de la proteína. La estabilidad de las proteínas está influida por diversas variables en el entorno de la solución, como el pH, el tipo de amortiguador, la fuerza iónica, los excipientes y los detergentes. Estos efectos pueden observarse como un pequeño cambio de la Tmáx o como cambios más grandes de hasta decenas de grados [6]. Diseñado teniendo en cuenta las necesidades de la industria biofarmacéutica, el rendimiento paralelo del RS-DSC de TA Instruments facilita el escaneo de las condiciones de la solución para fármacos proteína biológicos, lo que disminuye el tiempo hasta la toma de decisiones en comparación con los
métodos microcalorimétricos tradicionales.
Para demostrar cómo los datos del escaneo de la formulación pueden ayudar en la selección de los componentes del amortiguador, analizamos datos por triplicado del anticuerpo Herceptin Trastuzumab en cuatro condiciones comunes de amortiguador (figura 2 y tabla 1). Para este estudio, se escanearon anticuerpos en varios amortiguadores de formulación a ~ 20 mg/ml; sin embargo, el RS-DSC de TA Instruments está diseñado exclusivamente para manejar concentraciones de proteína de potencia de formulación de más de 330 mg/ml, lo que permite realizar pruebas de estabilidad térmica de medicamento no diluido. Para obtener más información, vea la nota de aplicación de TA Instruments “Escaneo rápido de estabilidad térmica de fármacos biológicos de alta concentración” (“Rapid Thermal Stability Screening of High Concentration Biologic Drugs“) [7].
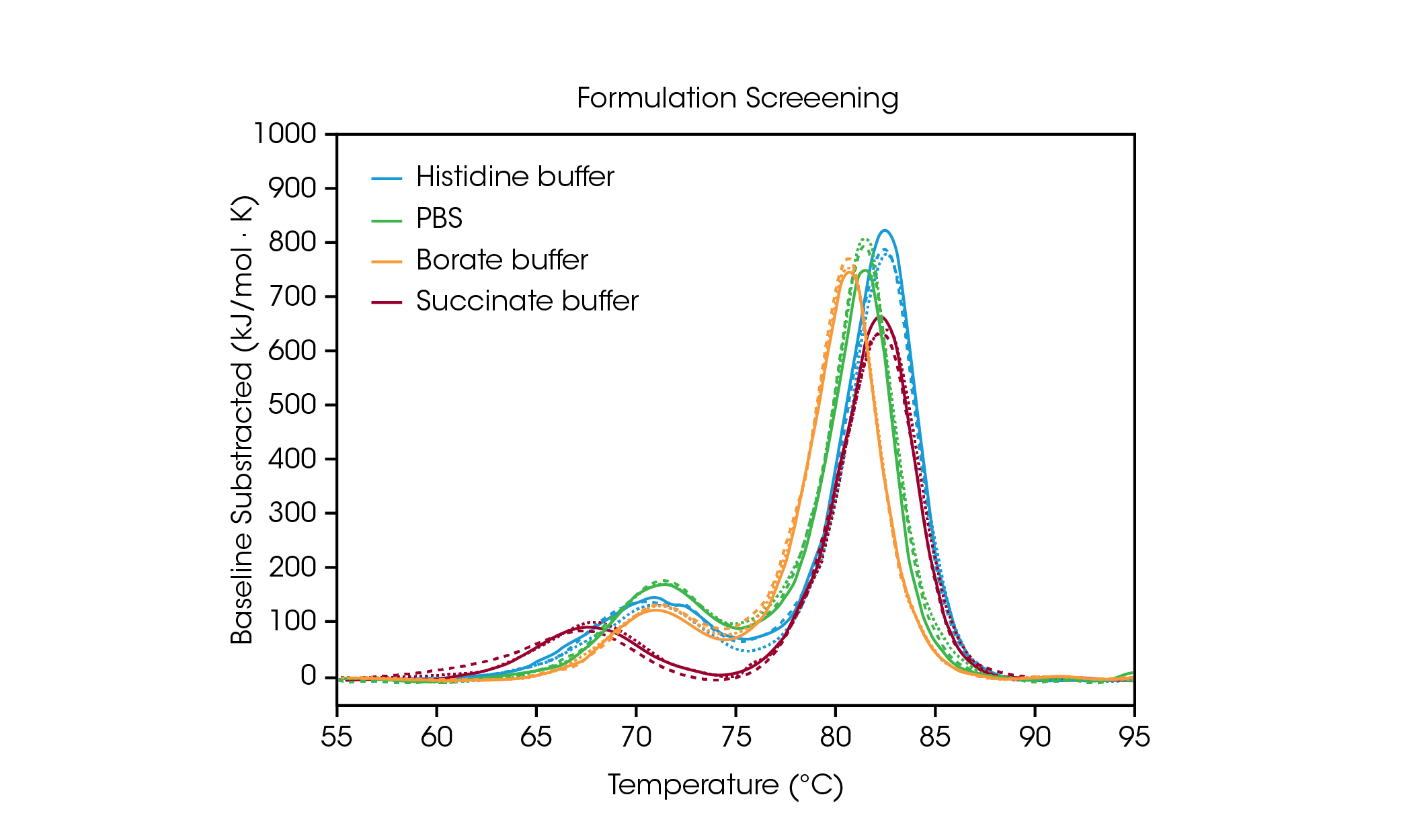
Tabla 1. Temperaturas de transición para el escaneo de formulación. Media ± DE, n = 3.
| Amortiguador | Tmáx,1 (°C) | Tmáx,2 (°C) |
|---|---|---|
| Histidina | 70.76 ± 0.38 | 82.66 ± 0.09 |
| Borato | 70.84 ± 0.08 | 80.69 ± 0.02 |
| PBS | 71.12 ± 0.06 | 81.48 ± 0.02 |
| Succinato | 67.71 ± 0.22 | 82.66 ± 0.08 |
Los amortiguadores histidina, borato o PBS no tienen repercusiones importantes sobre el primer evento de despliegue, que corresponde al despliegue del dominio CH2[8]. Sin embargo, el amortiguador succinato desestabiliza el dominio CH2 , lo que da por resultado disminución del inicio del despliegue y la Tmáx,1 aproximadamente 3 °C. Con respecto a la transición principal que refleja los eventos de despliegue de Fab y CH3 los amortiguadores de histidina y succinato son los más estabilizadores, con una Tmáx,2 de 82.66 °C. La transición principal es menos estable en el amortiguador de borato, con una Tmáx,2 de 80.69 °C. No sorprende que la formulación de amortiguador más estabilizadora para trastuzumab en este conjunto de muestras es el amortiguador de histidina que se utilizó para la formulación final del medicamento aprobado.
Para facilitar el análisis de datos del escaneo de la formulación, la detección automatizada de Tmáx en el software NanoAnalyze permite la detección precisa de hasta tres transiciones en escaneos de estabilidad de proteína. Para demostrar la precisión del algoritmo de selección de picos, Tmáx,1 y Tmáx,2 automáticos para trastuzumab a 50 mg/ml en amortiguador de formulación de histidina se comparan con la selección de picos manual y se describen en la Figura 3 y la Tabla 2. Para el análisis automatizado, se detectaron dos picos después de aplicar un polinomio basal de cuarto orden al utilizar la función de detección de gancho de inicio automático, que no requiere especificación del rango de temperatura de interés.
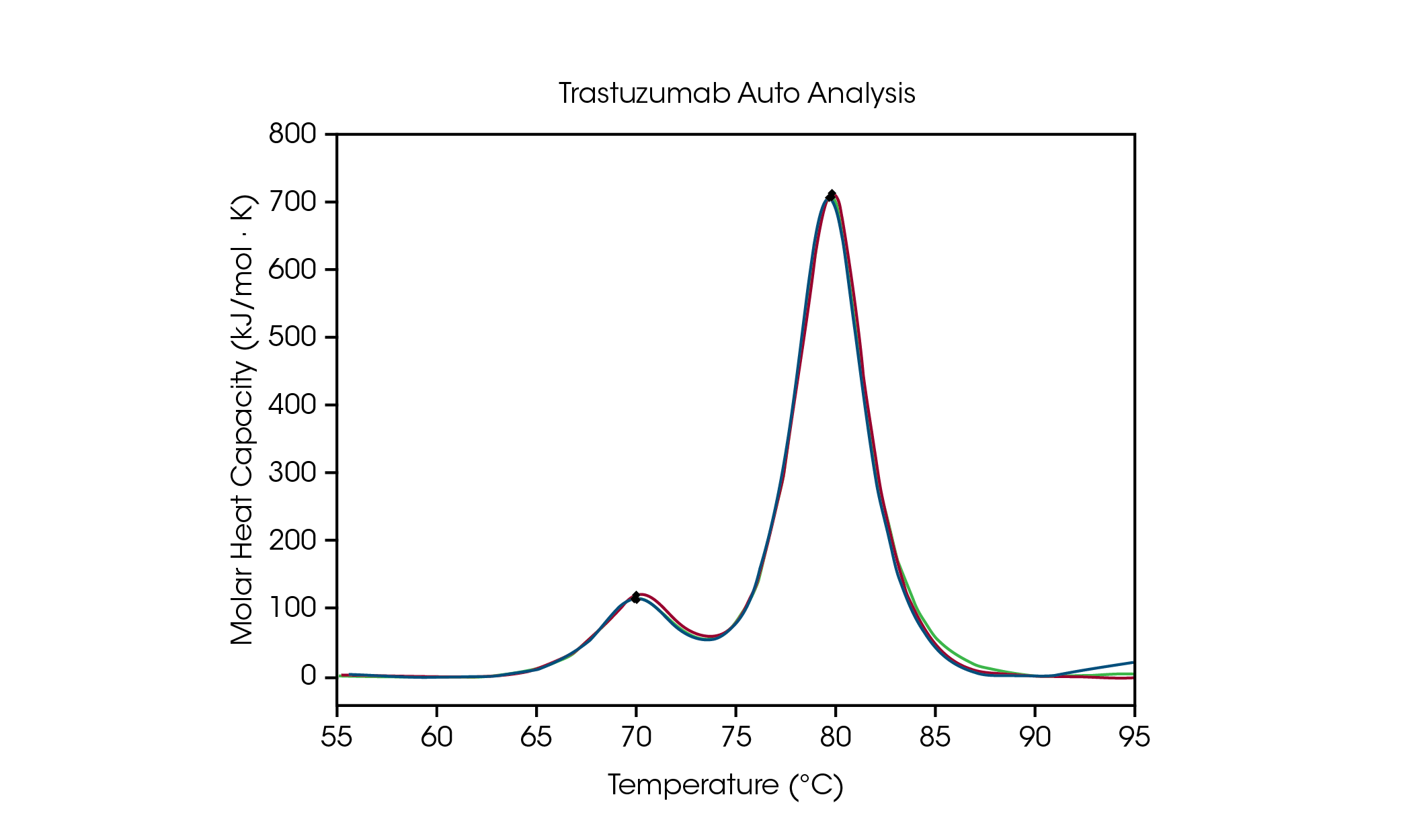
Tabla 2. Comparación entre la detección automatizada y manual de Tmáx para trastuzumab.
| Muestra | Tmáx,1 (°C) Automatizada | Tmáx,1 (°C) Manual | Tmáx,2 (°C) Automatizada | Tmáx,2 (°C) Manual |
|---|---|---|---|---|
| Réplica 1 | 71.93 | 72.04 | 82.92 | 82.88 |
| Réplica 2 | 71.81 | 71.93 | 82.76 | 82.75 |
| Réplica 3 | 71.93 | 71.97 | 82.90 | 82.83 |
| Promedio | 71.89 ± 0.07 | 71.98 ± 0.06 | 82.86 ± 0.09 | 82.82 ± 0.07 |
El despliegue del dominio CH2 tiene una Tmáx,1 automatizada de 71.89 °C en comparación con 71.98 °C para el análisis de picos manual. La transición principal tiene una Tmáx,2 automatizada de 82.86 °C en comparación con 82.82 °C para el análisis de picos manual. Se observa una excelente reproducibilidad para trastuzumab. Cuando se prueba por triplicado, se observa que la Tmáx de ambos picos está dentro de los límites de precisión del instrumento de ± 0.2 °C, al utilizar análisis de picos tanto manual como automatizado. Siempre se recomienda la inspección visual para asegurar una comprensión completa de los datos. Este conjunto de datos demuestra que las funciones de análisis de DSC automatizado proporcionan una detección precisa de Tmáx en termogramas de transición múltiple.
Escaneo de modificaciones de secuencia
Las mutaciones de proteína son una estrategia común para optimizar la estructura y función de la proteína, e incluso las modificaciones de un solo aminoácido pueden tener un efecto medible en la estabilidad general de la proteína. La DSC se utiliza para comprender las repercusiones de una mutación en la estabilidad térmica de una proteína, y puede ayudar a guiar la toma de decisiones en el proceso de desarrollo de fármacos biológicos. Para demostrar los tipos de efectos que una modificación de secuencia puede tener sobre la estabilidad, un pequeño panel de anticuerpos diseñados se escaneó para buscar cambios de despliegue térmico resultantes de una mutación de un solo aminoácido en la secuencia de la proteína (Figura 4 y Tabla 3).
En el anticuerpo original, el despliegue de los dominios CH2, Fab y CH3 está contenido dentro de una transición térmica principal a una Tmáx de 75.92 °C. A partir de la proteína original, se hizo una mutación de un solo aminoácido que no tiene efecto importante sobre la estabilidad térmica a corto plazo (Mutación 1) con una Tmax de 75.52 °C. Sin embargo, se ha demostrado que mutaciones adicionales de un solo aminoácido tienen repercusiones importantes en la estabilidad de la proteína (Mutación 2 y Mutación 3). En la Mutación 2 se observa un hombro evidente que probablemente corresponde a desestabilización en el dominio CH2 con una Tmáx,1 agregada de 70.04 °C junto con un pequeño cambio en la transición principal en comparación con la proteína original con una Tmáx,2 de 75.14 °C. La Mutación 3 causa una desestabilización significativa del dominio CH2, lo que da por resultado un pico de despliegue adicional completo con una Tmáx,1 de 64.85 °C. La transición principal relativamente no queda afectada con una Tmáx,2 de 75.29 °C. Como se ilustra en la importante desestabilización en la Mutación 3, la modificación no siempre tiene el mismo efecto; más bien, depende tanto del sitio de la modificación como de las propiedades fisicoquímicas del nuevo aminoácido. La optimización de los beneficios funcionales deseados de la modificación de secuencia con la estabilidad estructural de la proteína ayuda a comprender la relación estructura-función y puede facilitar el desarrollo de terapias avanzadas.
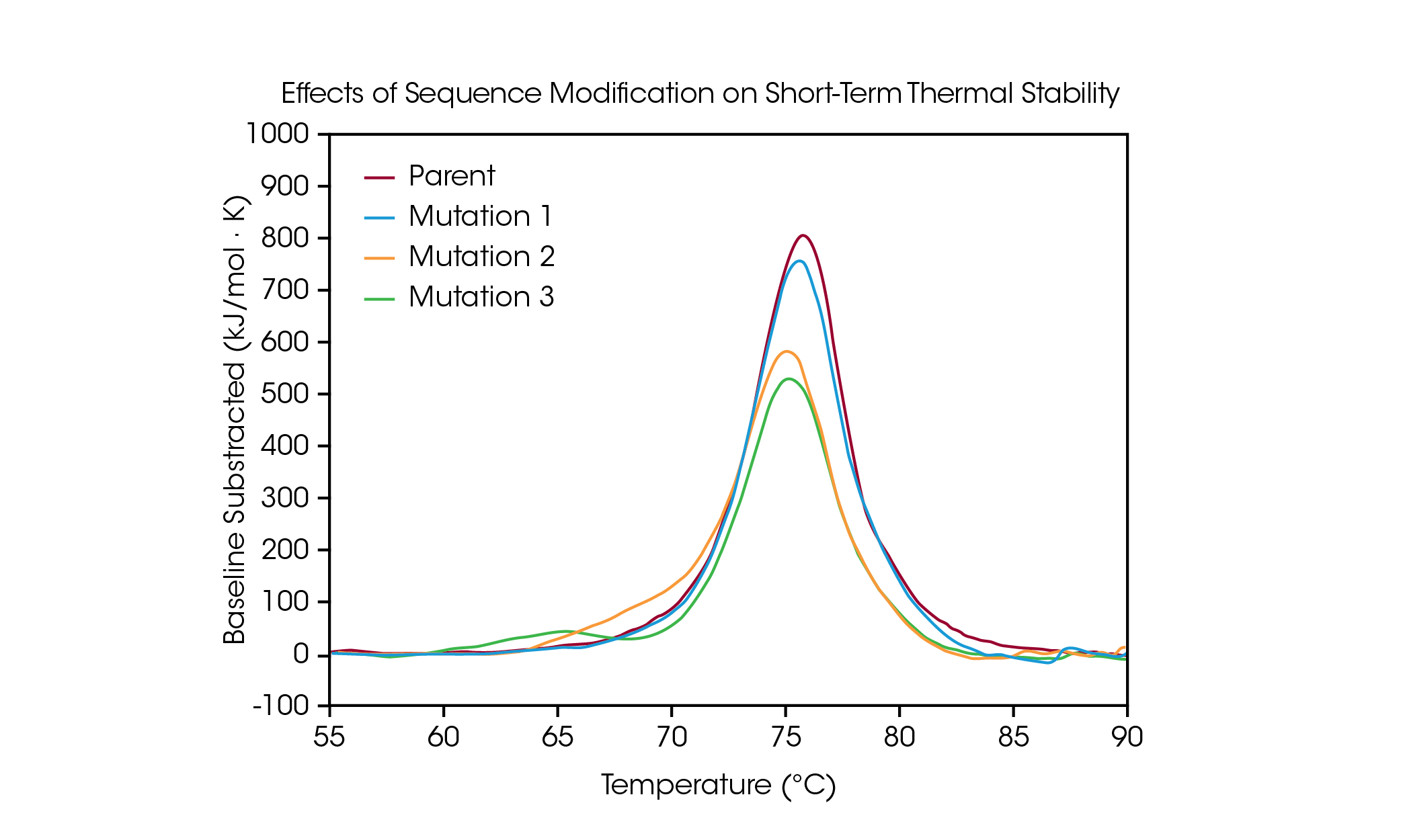
Tabla 3. Temperatura de transición para el escaneo de mutaciones. Media ± DE, n = 3. *Determinado mediante el modelo de Voigt.
| Muestra | Tmáx,1 (°C) | Tmáx,2 (°C) |
|---|---|---|
| Original | — | 75.92 ± 0.15 |
| Mutación 1 | — | 75.52 ± 0.14 |
| Mutación 2 | 70.04 ± 0.86* | 75.14 ± 0.06 |
| Mutación 3 | 64.85 ± 0.05* | 75.29 ± 0.06 |
Conclusiones
El escaneo de las repercusiones del entorno de la solución y la modificación de la secuencia sobre la estabilidad térmica a corto plazo es crucial para el desarrollo de productos farmacéuticos de alta calidad. El RS-DSC de TA Instruments permite un escaneo de alto rendimiento para la estabilidad térmica mediante pruebas simultáneas de hasta 24 muestras, lo que disminuye significativamente el tiempo hasta la toma de decisiones. En este documento demostramos un análisis preciso y reproducible de muestras de anticuerpos complejos-de transición múltiple y la determinación rápida del componente del amortiguador y las repercusiones de la mutación sobre la estructura general de la proteína. Los nuevos algoritmos en el software de procesamiento de datos, NanoAnalyze, facilitan el análisis de grandes cantidades de datos térmicos ahora accesibles con un escaneo de alto rendimiento. En general, el RS-DSC de TA Instruments proporciona una nueva plataforma para caracterizar la estabilidad de medicamentos con anticuerpos, una variable importante en la comprensión de la resiliencia al estrés térmico, la calidad del producto y el soporte de la aprobación regulatoria.
Referencias
- V. T. Oi, T. M. Vuong, R. Hardy, J. Reidler, J. Dangl, L. A. Herzenberg and L. Stryer, “Correlation between segmental flexibility and effector function of antibodies,” Nature, vol. 307, pp. 136-140, 1984.
- P. L. Privalov and S. A. Potekhin, “Scanning microcalorimetry in studying temperature-induced changes in proteins,” Methods in Enzymology, vol. 131, pp. 5-51, 1986.
- J. Wen, Y. Jiang and L. Nahri, “Effect of Carbohydrate on Thermal Stability of Antibodies,” American Pharmaceutical Review, vol. 11, p. 98, 2008.
- “Standard Practice for Calibration of Fixed-Cell Differential Scanning Calorimeters,” ASTM E2603-15, 2023.
- “NanoDSC: What to Consider when Choosing a Baseline and Model,” TA Instruments, MC154.
- C. M. Johnson, “Differential scanning calorimetry as a tool for protein folding and stability,” Archives of Biochemistry and Biophysics, vol. 531, pp. 100-109, 2013.
- “Rapid Thermal Stability Screening of High Concentration Biologic Drugs,” TA Instruments, MC177.
- K. J. Arlotta, A. V. Gandhi, H.-N. Chen, C. S. Nervig, J. F. Carpenter and S. C. Owen, “In-Depth Comparison of Lysine-Based Antibody-Drug Conjugates Prepared on Solid Support Versus in Solution,” Antibodies, vol. 7, p. 6, 2018.
Haga clic aquí para descargar la versión imprimible de esta nota de aplicación.

